«Folding@home»
So können Sie der Forschung im Kampf gegen das Coronavirus helfen
Das Projekt «Folding@home» nutzt die Rechenleistung von privaten Computern rund um die Welt, um die Struktur von Proteinen zu untersuchen. Neu dazugekommen sind nun die Proteine von SARS-CoV-2. So kann jeder mithelfen, sie zu erfoschen.
Um zu verstehen, wie ein Virus genau funktioniert, muss man sich seine Bausteine anschauen. Das sind – wie bei allen lebenden Organismen auf diesem Planeten auch – Proteine. Diese wiederum sind allerdings alles andere als einfache Gebilde. Sie bestehen im Grunde aus Ketten von Molekülen, die erst einmal richtig angeordnet sein müssen, damit überhaupt ein funktionelles Protein draus werden kann. Letztendlich ist die Postition jedes einzelnen Atoms in jedem einzelnen Molekül entscheidend.
Als nächstes müssen die Molekül-Ketten dann auf eine bestimmte Weise gefaltet werden. Viele Proteine bestehen zudem aus mehreren solchen gefalteten Ketten, die zusammen ein Gefüge bilden. Auch der Aufbau dieses Gefüges muss nach einem vorgegebenen Muster erfolgen. Zudem ist das durch chemische Bindungen zusammengehaltene Gefüge auch ständig in Bewegung.
Kennt man die Struktur des Gefüges, weiss man, wie die Moleküle angeordnet sind, kann man daraus sehr viel herauslesen. Beispielsweise kann so ermittelt werden, wo Medikamente am besten andocken müssen, damit sie das Protein zerstören und dafür sorgen können, dass ein Virus sich beispielsweise nicht mehr vermehren oder in die Zelle eintreten kann.
Allerdings ist die Rechenleistung eines Computers, die es braucht, um diese sogenannte Faltstruktur eines Proteins zu ermitteln, immens. Wie Computerbiophysiker Gregory Bowman in einem Video erklärt, bräuchte es für die Simulation eines Antibiotika-Resistenzproteins auf seinem MacBook Pro 500 Jahre.
Proteine zuhause falten
Findige Wissenschaftler der Standford University in Kalifornien fanden daher eine andere Lösung: Die Simulation einfach in kleinen Brocken auf ganz viele Rechner zu verteilen und dann wieder zusammenzufügen. Dieses Projekt nennt sich «Folding@home» (falten zuhause) und jeder, der einen Computer besitzt, kann mitmachen. Dazu lädt man sich einfach eine Software herunter, welche die Berechnungen im Hintergrund anstellt und die Ergebnisse zurück ins Labor sendet, während man selbst am Computer etwas anderes macht. Genutzt dazu wird freie Prozessorzeit.
«Folding@home» gibt es seit bald zwanzig Jahren und erforscht Krankheiten wie Krebs, Alzheimer, Parkinson oder Ebola – und seit einer Woche auch COVID-19. Wie Gregory Bowman, der Direktor des Projekts ist, in einem Blogpost schreibt, gehe es darum, die Dynamiken der Proteine des neuen Coronavirus zu simulieren, um nach neuen therapeutischen Möglichkeiten zu suchen. Bowman verspricht, die so generierten Daten schnellstmöglich der Wissenschaft zur Verfügung zu stellen, damit diese nach einem Medikament forschen kann.
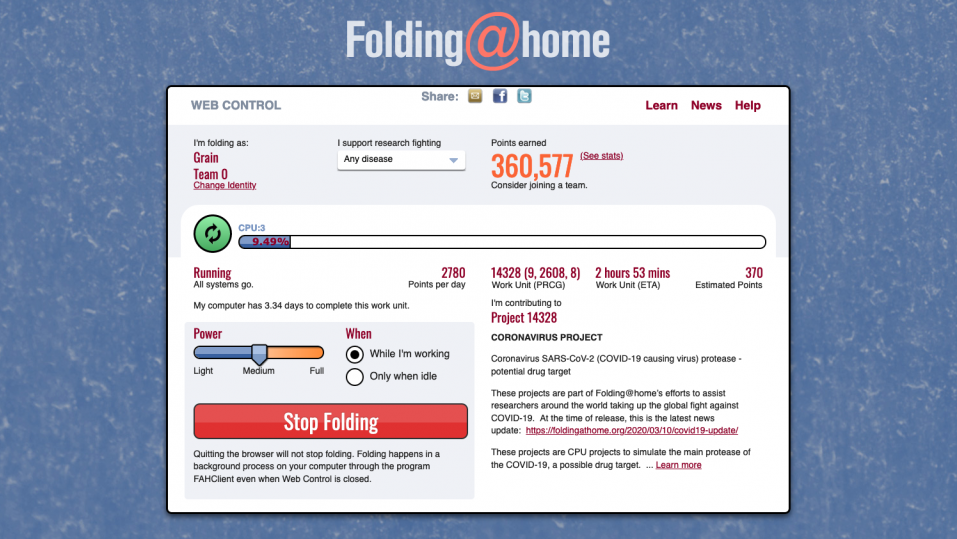
So installieren Sie «Folding@home» Laden Sie die Software für Ihr Gerät hier herunter. Die Seite sollte automatisch erkennen, ob sie einen Windows-PC oder einen Mac nutzen und Ihnen das Richtige zur Verfügung stellen. Folgen Sie den Anweisungen des Installierungs-Assistenten. Navigieren Sie auf Ihrem PC oder Mac zum Ordner «Folding@home». Starten Sie die Applikation «FAHControl» – an den Einstellungen dort müssen Sie nichts ändern, es sollte automatisch zu laufen beginnen. Eventuell kann es ein wenig dauern, bis ihr Computer einen Auftrag zum Falten bekommt. Falls sich Ihr Browser nicht schon nach der Installation geöffnet hat, öffnen Sie die Datei «Web Control.url». Es geht ein Browserfenster auf, das anzeigt, für welches Projekt Sie gerade Proteine falten. Unter «Change Identity» können Sie sich einen anderen Namen geben. Wenn Sie beim Drop-down-Menu unter «I support research fighting» «Any disease» anwählen, kommen Sie in das Corona-Projekt. Sobald Ihnen ein Faltauftrag zugeteilt wird, fängt der Computer automatisch an zu arbeiten. Sollte er etwas lauter tönen als sonst, müssen sie sich keine Sorgen machen. Er braucht zum Falten nur die Prozessorleistung, die sonst ungenutzt bliebe.
[IMG 2]
Dieser Artikel wurde automatisch auf unsere neue Website übertragen. Es kann daher sein, dass Darstellungsfehler auftreten. Diese können Sie uns mit folgendem Formular melden. Vielen Dank für Ihr Verständnis.













Bitte loggen Sie sich ein, um die Kommentarfunktion zu nutzen.
Falls Sie noch kein Agrarmedien-Login besitzen:
Jetzt registrieren